Capítulo6 Transformação
Aviso: Para ler a versão mais recente deste material, acesse: https://gustavojy.github.io/apostila-icdr/
A etapa de transformação dos dados consiste em selecionar as variáveis e observações de interesse no nosso banco de dados, a fim de gerar medidas úteis para a análise. Podemos realizar operações entre colunas de acordo com determinada variável, calcular a média, mediana, contagem e porcentagens, além de selecionar, filtrar e criar novas variáveis.
Os principais pacotes relacionados ao tema, presentes no tidyverse, são o dplyr, stringr, forcats e lubridate, cada qual apresentando funções particulares e específicas para trabalhar com os dados. Nesta apostila, abordaremos apenas o pacote dplyr, cujas funções conseguem resolver a maioria dos problemas relacionados a essa etapa.
Os demais pacotes tratam de assuntos específicos na transformação de dados. Caso você precise tratar de algum problema que o dplyr não consiga resolver, descreveremos, brevemente, as características desses pacotes para facilitar suas pesquisas.
stringr: manipula as variáveis categóricas a partir de expressões regulares (REGEX);forcats: apresenta funções que lidam com variáveis do tipo fator (factor). Caso queira entender melhor sobre esse tipo de classe, confira a seção 3.10;lubridate: pacote específico para trabalhar com variáveis do tipo data e tempo.
A seguir, trataremos com detalhes as funcionalidades presentes no pacote dplyr. Para tanto, precisamos rodar o pacote.
Para verificar todas as funcionalidades presentes no pacote, rode o seguinte comando:
6.1 Pacote dplyr
O pacote dplyr possui ferramentas simples, porém muito importantes para realizar as devidas transformações na base de dados. A seguir, apresentaremos as principais funções do pacote, que nos permitem resolver a maioria dos problemas relacionados à etapa de transformação dos dados.
A base de dados utilizada para os exemplos é referente à produção de milho, soja, trigo e arroz, nos países da América do Sul, entre 1961 e 2019, obtidos da FAOSTAT. Para fazer o download dos dados, clique aqui.
# A tibble: 5,510 × 14
Domain Cod…¹ Domain Area …² Area Eleme…³ Element Item …⁴ Item Year …⁵ Year
<chr> <chr> <dbl> <chr> <dbl> <chr> <dbl> <chr> <dbl> <dbl>
1 QCL Crops… 9 Arge… 5312 Area h… 56 Maize 1961 1961
2 QCL Crops… 9 Arge… 5312 Area h… 56 Maize 1962 1962
3 QCL Crops… 9 Arge… 5312 Area h… 56 Maize 1963 1963
4 QCL Crops… 9 Arge… 5312 Area h… 56 Maize 1964 1964
5 QCL Crops… 9 Arge… 5312 Area h… 56 Maize 1965 1965
6 QCL Crops… 9 Arge… 5312 Area h… 56 Maize 1966 1966
7 QCL Crops… 9 Arge… 5312 Area h… 56 Maize 1967 1967
8 QCL Crops… 9 Arge… 5312 Area h… 56 Maize 1968 1968
9 QCL Crops… 9 Arge… 5312 Area h… 56 Maize 1969 1969
10 QCL Crops… 9 Arge… 5312 Area h… 56 Maize 1970 1970
# … with 5,500 more rows, 4 more variables: Unit <chr>, Value <dbl>,
# Flag <chr>, `Flag Description` <chr>, and abbreviated variable names
# ¹`Domain Code`, ²`Area Code (FAO)`, ³`Element Code`, ⁴`Item Code (FAO)`,
# ⁵`Year Code`O banco de dados possui 5510 observações e 14 variáveis. Como podemos observar, muitas das variáveis são referentes à códigos de identificação, os quais não nos interessam para a realização das análises. Assim, a seguir, veremos como selecionar somente as variáveis de interesse.
6.1.1 Selecionar
Para selecionar colunas, utilizamos a função select(), tendo como primeiro argumento a base de dados utilizada, sendo os demais argumentos referentes aos nomes das colunas que se deseja selecionar.
Nos dois exemplos a seguir, perceba que podemos selecionar uma ou mais de uma coluna.
# A tibble: 5,510 × 1
Item
<chr>
1 Maize
2 Maize
3 Maize
4 Maize
5 Maize
6 Maize
7 Maize
8 Maize
9 Maize
10 Maize
# … with 5,500 more rows# A tibble: 5,510 × 3
Area Item Value
<chr> <chr> <dbl>
1 Argentina Maize 2744400
2 Argentina Maize 2756670
3 Argentina Maize 2645400
4 Argentina Maize 2970500
5 Argentina Maize 3062300
6 Argentina Maize 3274500
7 Argentina Maize 3450500
8 Argentina Maize 3377700
9 Argentina Maize 3556000
10 Argentina Maize 4017330
# … with 5,500 more rowsPodemos selecionar várias colunas consecutivas com o operador :. Basta informar os nomes ou as posições da primeira e da última coluna que se deseja selecionar.
# A tibble: 5,510 × 7
Area `Element Code` Element `Item Code (FAO)` Item Year …¹ Year
<chr> <dbl> <chr> <dbl> <chr> <dbl> <dbl>
1 Argentina 5312 Area harvested 56 Maize 1961 1961
2 Argentina 5312 Area harvested 56 Maize 1962 1962
3 Argentina 5312 Area harvested 56 Maize 1963 1963
4 Argentina 5312 Area harvested 56 Maize 1964 1964
5 Argentina 5312 Area harvested 56 Maize 1965 1965
6 Argentina 5312 Area harvested 56 Maize 1966 1966
7 Argentina 5312 Area harvested 56 Maize 1967 1967
8 Argentina 5312 Area harvested 56 Maize 1968 1968
9 Argentina 5312 Area harvested 56 Maize 1969 1969
10 Argentina 5312 Area harvested 56 Maize 1970 1970
# … with 5,500 more rows, and abbreviated variable name ¹`Year Code`# A tibble: 5,510 × 7
Area `Element Code` Element `Item Code (FAO)` Item Year …¹ Year
<chr> <dbl> <chr> <dbl> <chr> <dbl> <dbl>
1 Argentina 5312 Area harvested 56 Maize 1961 1961
2 Argentina 5312 Area harvested 56 Maize 1962 1962
3 Argentina 5312 Area harvested 56 Maize 1963 1963
4 Argentina 5312 Area harvested 56 Maize 1964 1964
5 Argentina 5312 Area harvested 56 Maize 1965 1965
6 Argentina 5312 Area harvested 56 Maize 1966 1966
7 Argentina 5312 Area harvested 56 Maize 1967 1967
8 Argentina 5312 Area harvested 56 Maize 1968 1968
9 Argentina 5312 Area harvested 56 Maize 1969 1969
10 Argentina 5312 Area harvested 56 Maize 1970 1970
# … with 5,500 more rows, and abbreviated variable name ¹`Year Code`A função select() possui outras funções que auxiliam na seleção de colunas, sendo elas:
starts_with(): seleciona colunas que começam com um texto padrão;ends_with(): seleciona colunas que terminam com um texto padrão;contains(): seleciona colunas que possuem um texto padrão.
# A tibble: 5,510 × 2
`Year Code` Year
<dbl> <dbl>
1 1961 1961
2 1962 1962
3 1963 1963
4 1964 1964
5 1965 1965
6 1966 1966
7 1967 1967
8 1968 1968
9 1969 1969
10 1970 1970
# … with 5,500 more rows# A tibble: 5,510 × 3
`Domain Code` `Element Code` `Year Code`
<chr> <dbl> <dbl>
1 QCL 5312 1961
2 QCL 5312 1962
3 QCL 5312 1963
4 QCL 5312 1964
5 QCL 5312 1965
6 QCL 5312 1966
7 QCL 5312 1967
8 QCL 5312 1968
9 QCL 5312 1969
10 QCL 5312 1970
# … with 5,500 more rows# A tibble: 5,510 × 2
`Area Code (FAO)` `Item Code (FAO)`
<dbl> <dbl>
1 9 56
2 9 56
3 9 56
4 9 56
5 9 56
6 9 56
7 9 56
8 9 56
9 9 56
10 9 56
# … with 5,500 more rowsTambém podemos retirar uma coluna inserindo um sinal de menos (-) antes do nome da variável ou das funções auxiliares.
[1] "Domain Code" "Domain" "Area Code (FAO)" "Area"
[5] "Element Code" "Element" "Item Code (FAO)" "Item"
[9] "Year Code" "Year" "Unit" "Value"
[13] "Flag" "Flag Description"# A tibble: 5,510 × 6
Area Element Item Year Unit Value
<chr> <chr> <chr> <dbl> <chr> <dbl>
1 Argentina Area harvested Maize 1961 ha 2744400
2 Argentina Area harvested Maize 1962 ha 2756670
3 Argentina Area harvested Maize 1963 ha 2645400
4 Argentina Area harvested Maize 1964 ha 2970500
5 Argentina Area harvested Maize 1965 ha 3062300
6 Argentina Area harvested Maize 1966 ha 3274500
7 Argentina Area harvested Maize 1967 ha 3450500
8 Argentina Area harvested Maize 1968 ha 3377700
9 Argentina Area harvested Maize 1969 ha 3556000
10 Argentina Area harvested Maize 1970 ha 4017330
# … with 5,500 more rowsPor último, temos a função everything(), utilizada na função select() para arrastar determinadas colunas para o início da base de dados.
# A tibble: 5,510 × 14
Value Unit Domain Cod…¹ Domain Area …² Area Eleme…³ Element Item …⁴ Item
<dbl> <chr> <chr> <chr> <dbl> <chr> <dbl> <chr> <dbl> <chr>
1 2744400 ha QCL Crops… 9 Arge… 5312 Area h… 56 Maize
2 2756670 ha QCL Crops… 9 Arge… 5312 Area h… 56 Maize
3 2645400 ha QCL Crops… 9 Arge… 5312 Area h… 56 Maize
4 2970500 ha QCL Crops… 9 Arge… 5312 Area h… 56 Maize
5 3062300 ha QCL Crops… 9 Arge… 5312 Area h… 56 Maize
6 3274500 ha QCL Crops… 9 Arge… 5312 Area h… 56 Maize
7 3450500 ha QCL Crops… 9 Arge… 5312 Area h… 56 Maize
8 3377700 ha QCL Crops… 9 Arge… 5312 Area h… 56 Maize
9 3556000 ha QCL Crops… 9 Arge… 5312 Area h… 56 Maize
10 4017330 ha QCL Crops… 9 Arge… 5312 Area h… 56 Maize
# … with 5,500 more rows, 4 more variables: `Year Code` <dbl>, Year <dbl>,
# Flag <chr>, `Flag Description` <chr>, and abbreviated variable names
# ¹`Domain Code`, ²`Area Code (FAO)`, ³`Element Code`, ⁴`Item Code (FAO)`6.1.2 Operador pipe (%>%)
Na maior parte dos casos, utilizaremos mais de uma função para manipular os nossos dados. Com isso, entra em cena o pipe (%>%). O pipe está presente no pacote magrittr, que está contido no tidyverse. Portanto, antes de aprofundarmos na ideia central do pipe, devemos carregar o pacote magrittr.
A principal função do pipe é conectar linhas de códigos que se relacionam, executando-as em sequência, de uma só vez. A essa estrutura de código chamamos de pipelines. Como exemplo hipotético, calcularemos a média final de um aluno na disciplina de cálculo II, arredondando-a com uma casa decimal.
[1] 7.7[1] 7.7Utilizando o pipe, evitamos de escrever funções dentro de funções, ordenando-as de acordo com a ordem em que desejamos realizar as operações. No exemplo, calculamos primeiro a média das notas e, posteriormente, arrendondamos.
Quando utilizamos o pipe, obtemos um código mais legível, claro e compacto, principalmente quando trabalhamos com diversas funções. Isso facilita não somente a leitura, mas também na manutenção do código, caso seja preciso realizar alterações ou consertar possíveis problemas.
Tendo essa noção básica do que é o pipe, começaremos a aplicá-lo na manipulação dos dados. Caso queira saber mais sobre o pipe, confira o capítulo 18 do livro R for Data Science.
6.1.3 Filtrar
Podemos filtrar determinados valores que estão contidos nas linhas de cada coluna, sejam eles quantitativos ou categóricos. Para isso, utilizamos testes lógicos dentro da função filter().
# A tibble: 459 × 14
Domain Cod…¹ Domain Area …² Area Eleme…³ Element Item …⁴ Item Year …⁵ Year
<chr> <chr> <dbl> <chr> <dbl> <chr> <dbl> <chr> <dbl> <dbl>
1 QCL Crops… 40 Chile 5312 Area h… 56 Maize 1961 1961
2 QCL Crops… 40 Chile 5312 Area h… 56 Maize 1962 1962
3 QCL Crops… 40 Chile 5312 Area h… 56 Maize 1963 1963
4 QCL Crops… 40 Chile 5312 Area h… 56 Maize 1964 1964
5 QCL Crops… 40 Chile 5312 Area h… 56 Maize 1965 1965
6 QCL Crops… 40 Chile 5312 Area h… 56 Maize 1966 1966
7 QCL Crops… 40 Chile 5312 Area h… 56 Maize 1967 1967
8 QCL Crops… 40 Chile 5312 Area h… 56 Maize 1968 1968
9 QCL Crops… 40 Chile 5312 Area h… 56 Maize 1969 1969
10 QCL Crops… 40 Chile 5312 Area h… 56 Maize 1970 1970
# … with 449 more rows, 4 more variables: Unit <chr>, Value <dbl>, Flag <chr>,
# `Flag Description` <chr>, and abbreviated variable names ¹`Domain Code`,
# ²`Area Code (FAO)`, ³`Element Code`, ⁴`Item Code (FAO)`, ⁵`Year Code`No exemplo anterior, filtramos a coluna Area para que nos retornasse somente as observações referentes ao país Chile. Para isso, utilizamos o teste lógico ==, ou seja, pedimos para que nos retornasse somente as observações que apresentem o valor igual a Chile na coluna Area.
Lembre que a função filter() segue a lógica demontrada na seção 3.11. Mas perceba que, diferentemente do que fora exposto na referente seção, a filter() é muito mais simples e intuitiva de ser utilizada.
Também podemos selecionar um conjunto de valores contidos em uma coluna. Para isso, criamos um vetor com os valores desejados e aplicamos o teste %in%, ou seja, dentro da coluna Area, pedimos para que nos retorne somente os valores que estão contidos no vetor.
conjunto_paises <- filter(graos,
Area %in% c("Brazil", "Argentina", "Chile"))
unique(conjunto_paises$Area)[1] "Argentina" "Brazil" "Chile" A função unique() comprova a seleção dos respectivos países filtrados, nos retornando todos os valores únicos contidos na coluna Area após a utilização do filtro.
Por outro lado, podemos retirar valores com o operador !.
retirando_paises <- filter(graos,
!(Area %in% c("Brazil", "Argentina", "Chile")))
unique(retirando_paises$Area) [1] "Bolivia (Plurinational State of)" "Colombia"
[3] "Ecuador" "French Guyana"
[5] "Guyana" "Paraguay"
[7] "Peru" "Suriname"
[9] "Uruguay" "Venezuela (Bolivarian Republic of)"Da mesma forma, podemos aplicar os filtros para variáveis quantitativas. Note no exemplo a seguir que aplicamos 3 filtros. O primeiro referente à variável categórica Element, sendo os outros dois, às variáveis quantitativas Year e Value.
# A tibble: 54 × 14
Domain Cod…¹ Domain Area …² Area Eleme…³ Element Item …⁴ Item Year …⁵ Year
<chr> <chr> <dbl> <chr> <dbl> <chr> <dbl> <chr> <dbl> <dbl>
1 QCL Crops… 9 Arge… 5510 Produc… 56 Maize 2011 2011
2 QCL Crops… 9 Arge… 5510 Produc… 56 Maize 2012 2012
3 QCL Crops… 9 Arge… 5510 Produc… 56 Maize 2013 2013
4 QCL Crops… 9 Arge… 5510 Produc… 56 Maize 2014 2014
5 QCL Crops… 9 Arge… 5510 Produc… 56 Maize 2015 2015
6 QCL Crops… 9 Arge… 5510 Produc… 56 Maize 2016 2016
7 QCL Crops… 9 Arge… 5510 Produc… 56 Maize 2017 2017
8 QCL Crops… 9 Arge… 5510 Produc… 56 Maize 2018 2018
9 QCL Crops… 9 Arge… 5510 Produc… 56 Maize 2019 2019
10 QCL Crops… 9 Arge… 5510 Produc… 236 Soyb… 2011 2011
# … with 44 more rows, 4 more variables: Unit <chr>, Value <dbl>, Flag <chr>,
# `Flag Description` <chr>, and abbreviated variable names ¹`Domain Code`,
# ²`Area Code (FAO)`, ³`Element Code`, ⁴`Item Code (FAO)`, ⁵`Year Code`Para melhorar a organização e a apresentação da base de dados, podemos aplicar as funções filter() e select() juntas. Para tanto, utilizaremos o pipe para mesclar ambas as funções em uma pipeline.
graos %>%
filter(Element == "Production",
Area %in% c("Brazil", "Argentina"),
Year > 2010) %>%
select(Area, Element, Item, Year, Unit, Value)# A tibble: 72 × 6
Area Element Item Year Unit Value
<chr> <chr> <chr> <dbl> <chr> <dbl>
1 Argentina Production Maize 2011 tonnes 23799830
2 Argentina Production Maize 2012 tonnes 21196637
3 Argentina Production Maize 2013 tonnes 32119211
4 Argentina Production Maize 2014 tonnes 33087165
5 Argentina Production Maize 2015 tonnes 33817744
6 Argentina Production Maize 2016 tonnes 39792854
7 Argentina Production Maize 2017 tonnes 49475895
8 Argentina Production Maize 2018 tonnes 43462323
9 Argentina Production Maize 2019 tonnes 56860704
10 Argentina Production Rice, paddy 2011 tonnes 1748075
# … with 62 more rowsPerceba que a aplicação do pipe é bem simples e intuitiva. Primeiramente, indicamos a base de dados a ser utilizada para realizar a filtragem e seleção - no caso, a base graos. Em seguida, escrevemos o %>% para conectar o banco de dados com a função filter(); nesse caso, filtramos apenas os valores iguais a "Production" na coluna Element, os países Brazil e Argentina na variável Area e os anos maiores que 2010. Novamente, escrevemos o %>% para aplicar a select() e selecionar as variáveis desejadas.
Note que não foi preciso indicar, como primeiro argumento das funções, qual a base de dados utilizada, pois essa foi especificada na primeira parte da pipeline. Além disso, a execução do código é realizada na ordem em que são escritos os comandos. Assim, caso desejarmos filtrar uma coluna e, posteriomente, retirá-la da seleção, devemos nos atentar à ordem dos comandos.
graos %>%
filter(Area == "Brazil",
Element == "Production",
Year %in% c(2015:2019)) %>%
select(Item, Year, Value)# A tibble: 20 × 3
Item Year Value
<chr> <dbl> <dbl>
1 Maize 2015 85283074
2 Maize 2016 64188314
3 Maize 2017 97910658
4 Maize 2018 82366531
5 Maize 2019 101138617
6 Rice, paddy 2015 12301201
7 Rice, paddy 2016 10622189
8 Rice, paddy 2017 12464766
9 Rice, paddy 2018 11808412
10 Rice, paddy 2019 10368611
11 Soybeans 2015 97464936
12 Soybeans 2016 96394820
13 Soybeans 2017 114732101
14 Soybeans 2018 117912450
15 Soybeans 2019 114269392
16 Wheat 2015 5508451
17 Wheat 2016 6834421
18 Wheat 2017 4342812
19 Wheat 2018 5469236
20 Wheat 2019 5604158No exemplo acima, perceba que filtramos as colunas Area e Element, mas não às selecionamos posteriormente. Caso fosse realizada a seleção antes da filtragem, não seria possível filtrar as devidas variáveis, uma vez que não selecionamos suas colunas para, posteriormente, serem filtradas.
6.1.4 Modificar e criar colunas
Para modificar ou criar novas colunas, utilizamos a função mutate(). No exemplo a seguir, transformaremos os valores de produção, em toneladas, para quilogramas.
graos %>%
filter(Element == "Production") %>%
select(Area, Element, Item, Year, Value) %>%
mutate(Value = Value*1000)# A tibble: 2,756 × 5
Area Element Item Year Value
<chr> <chr> <chr> <dbl> <dbl>
1 Argentina Production Maize 1961 4850000000
2 Argentina Production Maize 1962 5220000000
3 Argentina Production Maize 1963 4360000000
4 Argentina Production Maize 1964 5350000000
5 Argentina Production Maize 1965 5140000000
6 Argentina Production Maize 1966 7040000000
7 Argentina Production Maize 1967 8510000000
8 Argentina Production Maize 1968 6560000000
9 Argentina Production Maize 1969 6860000000
10 Argentina Production Maize 1970 9360000000
# … with 2,746 more rowsNo código acima, transformamos os valores da coluna Value. Contudo, também podemos manter a coluna original e criar uma nova coluna com a variável calculada. Basta designar um novo nome à coluna, nesse caso, criamos a Value(kg).
graos %>%
filter(Element == "Production") %>%
select(Area, Element, Item, Year, Value) %>%
mutate(Value_kg = Value*1000)# A tibble: 2,756 × 6
Area Element Item Year Value Value_kg
<chr> <chr> <chr> <dbl> <dbl> <dbl>
1 Argentina Production Maize 1961 4850000 4850000000
2 Argentina Production Maize 1962 5220000 5220000000
3 Argentina Production Maize 1963 4360000 4360000000
4 Argentina Production Maize 1964 5350000 5350000000
5 Argentina Production Maize 1965 5140000 5140000000
6 Argentina Production Maize 1966 7040000 7040000000
7 Argentina Production Maize 1967 8510000 8510000000
8 Argentina Production Maize 1968 6560000 6560000000
9 Argentina Production Maize 1969 6860000 6860000000
10 Argentina Production Maize 1970 9360000 9360000000
# … with 2,746 more rowsPodemos realizar qualquer operação com a quantidade de colunas que desejarmos. Porém, deve ser retornado um vetor com comprimento igual à quantidade de linhas da base de dados ou com comprimento igual a 1, sendo assim realizado o processo de reciclagem do valor.
graos %>%
filter(Element == "Production") %>%
select(Area, Element, Item, Year, Value) %>%
mutate(Value = Value*1000,
Unit = "kg")# A tibble: 2,756 × 6
Area Element Item Year Value Unit
<chr> <chr> <chr> <dbl> <dbl> <chr>
1 Argentina Production Maize 1961 4850000000 kg
2 Argentina Production Maize 1962 5220000000 kg
3 Argentina Production Maize 1963 4360000000 kg
4 Argentina Production Maize 1964 5350000000 kg
5 Argentina Production Maize 1965 5140000000 kg
6 Argentina Production Maize 1966 7040000000 kg
7 Argentina Production Maize 1967 8510000000 kg
8 Argentina Production Maize 1968 6560000000 kg
9 Argentina Production Maize 1969 6860000000 kg
10 Argentina Production Maize 1970 9360000000 kg
# … with 2,746 more rows6.1.5 Resumo de valores
O processo de sumarizar consiste em resumir um conjunto de dados a partir de uma medida de interesse. Como exemplo, podemos tirar a média, mediana, frequência e proporção dos valores desejados. Para isso, utilizamos a função summarise(). A seguir, faremos a média da produção de milho no Brasil.
graos %>%
filter(Area == "Brazil",
Element == "Production",
Item == "Maize") %>%
select(Area, Item, Year, Value) %>%
summarise(media = mean(Value, na.rm = TRUE))# A tibble: 1 × 1
media
<dbl>
1 33776948.Podemos calcular várias medidas diferentes na função summarise().
graos %>%
filter(Area == "Brazil",
Element == "Production",
Item == "Maize") %>%
select(Area, Item, Year, Value) %>%
summarise(media = mean(Value),
mediana = median(Value),
variancia = var(Value))# A tibble: 1 × 3
media mediana variancia
<dbl> <dbl> <dbl>
1 33776948. 26589870 5.61e14Há casos em que queremos sumarizar uma coluna de acordo com alguma variável categórica de uma outra coluna. Para isso, utilizamos a função group_by() para indicar qual coluna desejamos agrupar para realizar a summarise(). No exemplo a seguir, agruparemos a variável Element para calcular a média da produção (Production) e da área colhida (Area harvested) de soja, na América do Sul.
graos %>%
filter(Item == "Soybeans") %>%
group_by(Element) %>%
summarise(media = mean(Value, na.rm = TRUE))# A tibble: 2 × 2
Element media
<chr> <dbl>
1 Area harvested 2132705.
2 Production 5335899.Podemos agrupar mais de duas variáveis para sumarizar. A seguir, agruparemos as colunas Area e Element para calcular, novamente, a média da produção e da área colhida, mas agora, por país sul-americano.
graos %>%
filter(Item == "Soybeans") %>%
group_by(Area, Element) %>%
summarise(media = mean(Value, na.rm = TRUE))# A tibble: 26 × 3
# Groups: Area [13]
Area Element media
<chr> <chr> <dbl>
1 Argentina Area harvested 6967484.
2 Argentina Production 17941231.
3 Bolivia (Plurinational State of) Area harvested 472537.
4 Bolivia (Plurinational State of) Production 922200.
5 Brazil Area harvested 12772243.
6 Brazil Production 32075459.
7 Chile Area harvested 984
8 Chile Production 1043.
9 Colombia Area harvested 45942.
10 Colombia Production 93380.
# … with 16 more rows6.1.6 Ordenar
Podemos ordenar as linhas da base de dados de acordo com algum parâmetro referente aos valores de uma ou mais colunas. Para tanto, utilizamos a função arrange().
# A tibble: 2,756 × 4
Area Item Year Value
<chr> <chr> <dbl> <dbl>
1 Chile Soybeans 1988 0
2 Chile Soybeans 1990 0
3 Chile Soybeans 1991 0
4 Chile Soybeans 1992 0
5 French Guyana Maize 1990 0
6 French Guyana Soybeans 1990 0
7 French Guyana Soybeans 1991 0
8 French Guyana Soybeans 1992 0
9 Guyana Soybeans 1990 0
10 Guyana Soybeans 1991 0
# … with 2,746 more rowsPor padrão, a função arrange() ordena os valores em ordem crescente. Para ordená-las em ordem decrescente, utilizamos a função desc() dentro da própria arrange().
graos %>%
filter(Element == "Production") %>%
select(Area, Item, Year, Value) %>%
arrange(desc(Value))# A tibble: 2,756 × 4
Area Item Year Value
<chr> <chr> <dbl> <dbl>
1 Brazil Soybeans 2018 117912450
2 Brazil Soybeans 2017 114732101
3 Brazil Soybeans 2019 114269392
4 Brazil Maize 2019 101138617
5 Brazil Maize 2017 97910658
6 Brazil Soybeans 2015 97464936
7 Brazil Soybeans 2016 96394820
8 Brazil Soybeans 2014 86760520
9 Brazil Maize 2015 85283074
10 Brazil Maize 2018 82366531
# … with 2,746 more rowsAlém disso, podemos ordenar a base de dados de acordo com duas variáveis.
graos %>%
filter(Element == "Production",
Item == "Rice, paddy") %>%
select(Area, Year, Value) %>%
arrange(Year, desc(Value))# A tibble: 754 × 3
Area Year Value
<chr> <dbl> <dbl>
1 Brazil 1961 5392477
2 Colombia 1961 473600
3 Peru 1961 331877
4 Guyana 1961 215103
5 Ecuador 1961 203000
6 Argentina 1961 149000
7 Chile 1961 104720
8 Venezuela (Bolivarian Republic of) 1961 80658
9 Suriname 1961 71562
10 Uruguay 1961 60866
# … with 744 more rowsPerceba que a ordem da declaração das variáveis na função arrange() altera a prioridade da ordenação.
graos %>%
filter(Element == "Production",
Item == "Rice, paddy") %>%
select(Area, Year, Value) %>%
arrange(desc(Value), Year)# A tibble: 754 × 3
Area Year Value
<chr> <dbl> <dbl>
1 Brazil 2011 13476994
2 Brazil 2004 13277008
3 Brazil 2005 13192863
4 Brazil 2009 12651144
5 Brazil 2017 12464766
6 Brazil 2015 12301201
7 Brazil 2014 12175602
8 Brazil 2008 12061465
9 Brazil 2018 11808412
10 Brazil 1988 11806450
# … with 744 more rowsNesse último exemplo, priorizamos a ordenação pelos valores de produção, em quanto que no outro, ordenamos a base a partir dos anos e, posteriormente, dos valores de produção.
6.1.7 Mudar nomes de colunas
Podemos alterar os nomes das colunas com a função rename(). Basta inserir o nome desejado e indicar, após o sinal de =, qual coluna da base de dados se deseja alterar o nome.
graos %>%
select(Area, Element, Item, Year, Unit, Value) %>%
rename(`país` = Area, tipo = Element, cultura = Item, ano = Year,
unidade = Unit, valor = Value)# A tibble: 5,510 × 6
país tipo cultura ano unidade valor
<chr> <chr> <chr> <dbl> <chr> <dbl>
1 Argentina Area harvested Maize 1961 ha 2744400
2 Argentina Area harvested Maize 1962 ha 2756670
3 Argentina Area harvested Maize 1963 ha 2645400
4 Argentina Area harvested Maize 1964 ha 2970500
5 Argentina Area harvested Maize 1965 ha 3062300
6 Argentina Area harvested Maize 1966 ha 3274500
7 Argentina Area harvested Maize 1967 ha 3450500
8 Argentina Area harvested Maize 1968 ha 3377700
9 Argentina Area harvested Maize 1969 ha 3556000
10 Argentina Area harvested Maize 1970 ha 4017330
# … with 5,500 more rows6.1.8 Juntar bases de dados
Em alguns casos, precisamos utilizar informações presentes em diferentes bases de dados, como por exemplo em planilhas Excel distintas ou em diferentes abas de uma mesma planilha. Nesse caso, é necessário juntar todas as informações em um único data frame.
Para isso, podemos utilizar algumas funções presentes no pacote dplyr. A seguir, apresentaremos as funções do tipo bind_ e _join, usando como exemplo a planilha dados_juntar.xlsx, presente no mesmo link para download apresentado no início desse capítulo.
[1] "dados1" "dados2" "dados3" "dados4" "dados5"Com a função readxl::excel_sheets(), verificamos que a planilha contém 5 abas. Essas abas tratam de um mesmo tema, as quais precisaremos juntar em um único data frame. Assim, devemos salvar cada aba da planilha em um objeto.
d1 <- read_excel("dados_transf/dados_juntar.xlsx", sheet = "dados1")
d2 <- read_excel("dados_transf/dados_juntar.xlsx", sheet = "dados2")
d3 <- read_excel("dados_transf/dados_juntar.xlsx", sheet = "dados3")
d4 <- read_excel("dados_transf/dados_juntar.xlsx", sheet = "dados4")
d5 <- read_excel("dados_transf/dados_juntar.xlsx", sheet = "dados5")Funções bind_
As funções do tipo bind_ são as mais simples para juntarmos os bancos de dados. A função bind_rows() junta as observações (linhas) de dois ou mais bancos de dados com base nas colunas. Com essa função, podemos realizar a união dos data frames d1, d2, d3 e d4, que possuem as mesmas variáveis (colunas), porém tratando de diferentes observações.
# A tibble: 2 × 4
nome sexo idade peso
<chr> <chr> <dbl> <dbl>
1 Amanda F 25 63
2 Maria F 30 65# A tibble: 2 × 4
nome sexo idade peso
<chr> <chr> <dbl> <dbl>
1 Vitor M 24 73
2 Leticia F 23 52# A tibble: 2 × 4
sexo nome peso idade
<chr> <chr> <dbl> <dbl>
1 F Vitoria 51 21
2 M Marcos 68 18# A tibble: 2 × 4
nomes sexo idade peso
<chr> <chr> <dbl> <dbl>
1 Julio M 25 72
2 Fabio M 35 81Primeiramente, uniremos as bases d1 e d2. Dentro da função bind_rows() declaramos os data frames que desejamos juntar, separados por vírgula.
# A tibble: 4 × 4
nome sexo idade peso
<chr> <chr> <dbl> <dbl>
1 Amanda F 25 63
2 Maria F 30 65
3 Vitor M 24 73
4 Leticia F 23 52Uma vez unidos os dados de d1 e d2, juntaremos com o d3. Porém, note que as colunas da base d3 apresentam uma ordem diferente em relação ao d1 e ao d2. Mesmo com a ordem diferente, a função consegue combinar as linhas de maneira adequada, desde que os nomes das colunas dos bancos de dados sejam iguais.
# A tibble: 6 × 4
nome sexo idade peso
<chr> <chr> <dbl> <dbl>
1 Amanda F 25 63
2 Maria F 30 65
3 Vitor M 24 73
4 Leticia F 23 52
5 Vitoria F 21 51
6 Marcos M 18 68A função bind_rows() consegue juntar mais de dois bancos de dados em um só comando. Basta declararmos as bases que desejamos juntar.
# Juntando d1, d2 e d3 em um só comando
juntar.linhas.direto <- bind_rows(d1, d2, d3)
juntar.linhas.direto# A tibble: 6 × 4
nome sexo idade peso
<chr> <chr> <dbl> <dbl>
1 Amanda F 25 63
2 Maria F 30 65
3 Vitor M 24 73
4 Leticia F 23 52
5 Vitoria F 21 51
6 Marcos M 18 68Por último, precisamos unir o d4 aos demais dados.
# A tibble: 8 × 5
nome sexo idade peso nomes
<chr> <chr> <dbl> <dbl> <chr>
1 Amanda F 25 63 <NA>
2 Maria F 30 65 <NA>
3 Vitor M 24 73 <NA>
4 Leticia F 23 52 <NA>
5 Vitoria F 21 51 <NA>
6 Marcos M 18 68 <NA>
7 <NA> M 25 72 Julio
8 <NA> M 35 81 FabioJuntando a base de dados d4 ao d1, d2 e d3, percebemos que foi criada uma nova coluna. Isso ocorre, pois a nomenclatura atribuída à variável nome no d4 está no plural, sendo assim, precisamos padronizar o nome dessa variável antes de juntá-la às demais bases.
[1] "nome" "sexo" "idade" "peso" # A tibble: 8 × 4
nome sexo idade peso
<chr> <chr> <dbl> <dbl>
1 Amanda F 25 63
2 Maria F 30 65
3 Vitor M 24 73
4 Leticia F 23 52
5 Vitoria F 21 51
6 Marcos M 18 68
7 Julio M 25 72
8 Fabio M 35 81Com a função rename(), renomeamos a coluna nomes para o singular (nome). Assim, ao realizar a junção dos quatro bancos de dados, os nomes são alocados em uma única coluna (nome).
Já a função bind_cols() une colunas de dois ou mais bancos de dados. A seguir, juntaremos as colunas das bases juntar.linhas e d5.
# A tibble: 8 × 4
nome sexo idade peso
<chr> <chr> <dbl> <dbl>
1 Amanda F 25 63
2 Maria F 30 65
3 Vitor M 24 73
4 Leticia F 23 52
5 Vitoria F 21 51
6 Marcos M 18 68
7 Julio M 25 72
8 Fabio M 35 81# A tibble: 8 × 2
nome profissao
<chr> <chr>
1 Fabio Medico
2 Gabriel Estudante
3 Guilherme Cozinheiro
4 Jose Biologo
5 Julio Zootecnista
6 Marcos Professor
7 Vitor Agronomo
8 Getulio Musico # A tibble: 8 × 6
nome...1 sexo idade peso nome...5 profissao
<chr> <chr> <dbl> <dbl> <chr> <chr>
1 Amanda F 25 63 Fabio Medico
2 Maria F 30 65 Gabriel Estudante
3 Vitor M 24 73 Guilherme Cozinheiro
4 Leticia F 23 52 Jose Biologo
5 Vitoria F 21 51 Julio Zootecnista
6 Marcos M 18 68 Marcos Professor
7 Julio M 25 72 Vitor Agronomo
8 Fabio M 35 81 Getulio Musico Note que a função juntou as colunas, preservando todas as variáveis, bem como a ordem original das linhas das bases de dados. Porém, nesse caso, seria conveniente unir as colunas com base na variável em comum entre os conjuntos, no caso, a variável nome. Para isso, utilizamos um outro conjunto de funções, as do tipo _join.
Funções _join
Para juntar dois conjuntos de dados com base em uma ou mais colunas em comum, utilizamos as funções do tipo _join (conhecidas também por merge). Você deve ter notado que a base d5 possui indivíduos em comum com a base juntar.linhas; mas também, diferentes.
Nesse caso, de acordo com o que se deseja para a análise, podemos selecionar todas as observações dos conjuntos; apenas as observações exclusivas de um dos conjuntos de dados; ou aquelas que estão presentes em ambas as bases de dados. A figura 6.1 ilustra tais possibilidades e as respectivas funções.
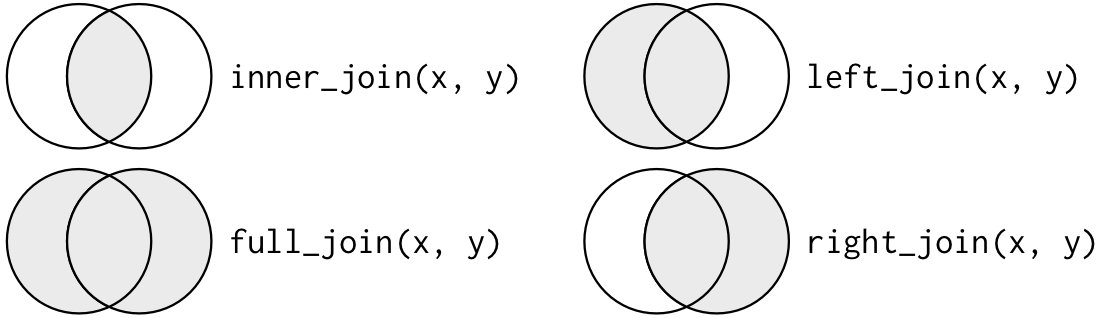
Figure 6.1: Diagrama de Venn com os tipos de joins. Fonte: R for Data Science, 2017.
A função inner_join() retorna as observações em comum entre os dois conjuntos, de acordo com certa variável.
# A tibble: 4 × 5
nome sexo idade peso profissao
<chr> <chr> <dbl> <dbl> <chr>
1 Vitor M 24 73 Agronomo
2 Marcos M 18 68 Professor
3 Julio M 25 72 Zootecnista
4 Fabio M 35 81 Medico Na função acima, declaramos as duas bases de dados a serem unidas, junto ao argumento by = "nome", sendo essa a variável em comum. Como resultado, a inner_join() nos retornou apenas a observações em comum entre o conjunto.
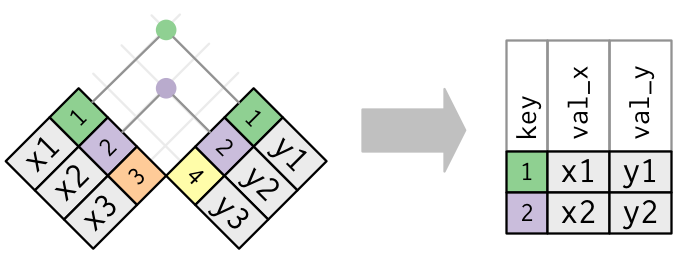
Figure 6.2: Esquematização da função inner-join. Fonte: R for Data Science, 2017.
Caso haja mais de uma variável em comum, bastaria declarar um vetor com as variáveis no argumento by = (by = c("var_1", "var_2", ..., "var_n")). Atente-se ao fato que as variáveis devem estar entre aspas.
A função full_join() nos retorna todas as observações de ambos os conjuntos, atribuindo valor NA para os valores ausentes.
# A tibble: 12 × 5
nome sexo idade peso profissao
<chr> <chr> <dbl> <dbl> <chr>
1 Amanda F 25 63 <NA>
2 Maria F 30 65 <NA>
3 Vitor M 24 73 Agronomo
4 Leticia F 23 52 <NA>
5 Vitoria F 21 51 <NA>
6 Marcos M 18 68 Professor
7 Julio M 25 72 Zootecnista
8 Fabio M 35 81 Medico
9 Gabriel <NA> NA NA Estudante
10 Guilherme <NA> NA NA Cozinheiro
11 Jose <NA> NA NA Biologo
12 Getulio <NA> NA NA Musico 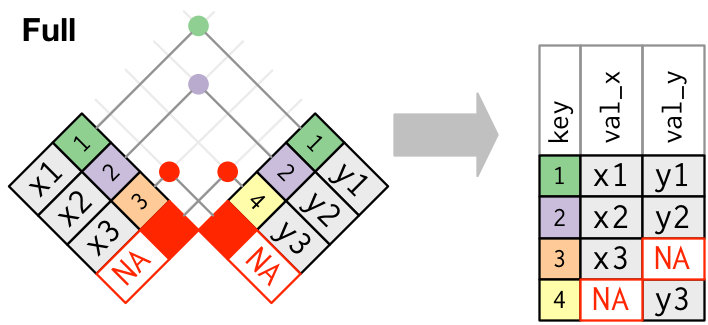
Figure 6.3: Esquematização da função full-join. Fonte: R for Data Science, 2017.
A função left_join() retorna todas as observações presentes no primeiro conjunto declarado, além dos valores em comum do segundo conjunto em relação ao primeiro. Por outro lado, a right_join() retorna todas as observações do segundo conjunto declarado e os valores em comum do primeiro conjunto em relação ao segundo.
# A tibble: 8 × 5
nome sexo idade peso profissao
<chr> <chr> <dbl> <dbl> <chr>
1 Amanda F 25 63 <NA>
2 Maria F 30 65 <NA>
3 Vitor M 24 73 Agronomo
4 Leticia F 23 52 <NA>
5 Vitoria F 21 51 <NA>
6 Marcos M 18 68 Professor
7 Julio M 25 72 Zootecnista
8 Fabio M 35 81 Medico # A tibble: 8 × 5
nome sexo idade peso profissao
<chr> <chr> <dbl> <dbl> <chr>
1 Vitor M 24 73 Agronomo
2 Marcos M 18 68 Professor
3 Julio M 25 72 Zootecnista
4 Fabio M 35 81 Medico
5 Gabriel <NA> NA NA Estudante
6 Guilherme <NA> NA NA Cozinheiro
7 Jose <NA> NA NA Biologo
8 Getulio <NA> NA NA Musico Note que em ambas as funções, o primeiro conjunto declarado é o juntar.linhas, sendo o segundo, o d5.
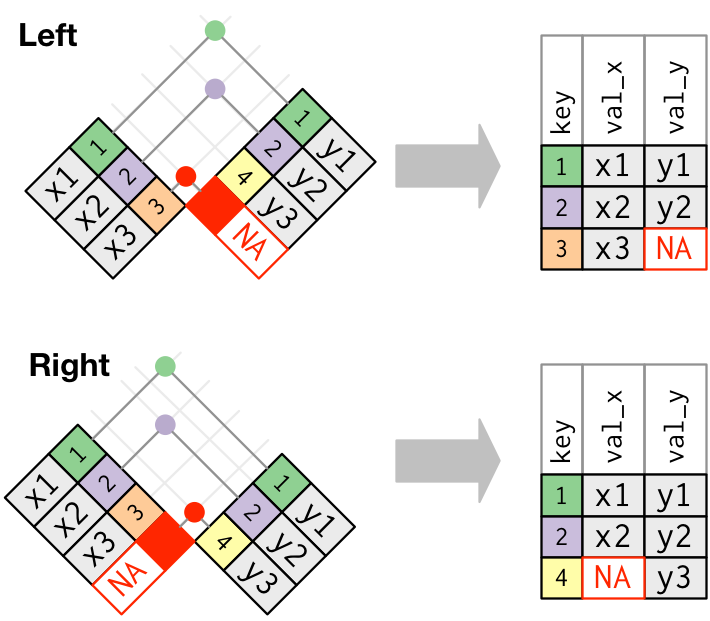
Figure 6.4: Esquematização das funções left-join e right-join. Fonte: R for Data Science, 2017.
Por fim, temos o semi_join() e o anti_join().
O semi_join() nos retorna todas as observações do primeiro conjunto que também estão presentes no segundo.
# Tendo como primeiro conjunto o `juntar.linhas`
semi.join1 <- semi_join(juntar.linhas, d5, by = "nome")
semi.join1# A tibble: 4 × 4
nome sexo idade peso
<chr> <chr> <dbl> <dbl>
1 Vitor M 24 73
2 Marcos M 18 68
3 Julio M 25 72
4 Fabio M 35 81# Tendo como primeiro conjunto o `d5`
semi.join2 <- semi_join(d5, juntar.linhas, by = "nome")
semi.join2# A tibble: 4 × 2
nome profissao
<chr> <chr>
1 Fabio Medico
2 Julio Zootecnista
3 Marcos Professor
4 Vitor Agronomo 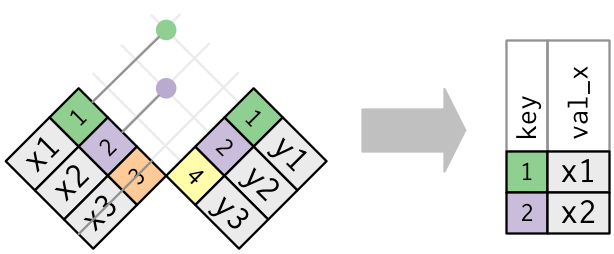
Figure 6.5: Esquematização da função semi-join. Fonte: R for Data Science, 2017.
Já o anti_join(), retorna as observações do primeiro conjunto que não estão presentes no segundo.
# Tendo como primeiro conjunto o `juntar.linhas`
anti.join1 <- anti_join(juntar.linhas, d5, by = "nome")
anti.join1# A tibble: 4 × 4
nome sexo idade peso
<chr> <chr> <dbl> <dbl>
1 Amanda F 25 63
2 Maria F 30 65
3 Leticia F 23 52
4 Vitoria F 21 51# Tendo como primeiro conjunto o `d5`
anti.join2 <- anti_join(d5, juntar.linhas, by = "nome")
anti.join2# A tibble: 4 × 2
nome profissao
<chr> <chr>
1 Gabriel Estudante
2 Guilherme Cozinheiro
3 Jose Biologo
4 Getulio Musico 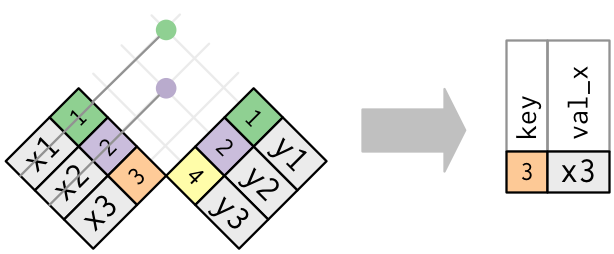
Figure 6.6: Esquematização da função anti-join. Fonte: R for Data Science, 2017.
Tanto no semi_join(), como no anti_join(), mantêm-se apenas as colunas presentes no primeiro conjunto.
Pode-se notar que o inner_join(), full_join(), left_join() e right_join() adicionam novas variáveis a um conjunto de dados a partir de observações correspondentes em outro, ou seja, são mutating joins (atuam de maneira semelhante à função mutate()). Já o semi_join() e o anti_join(), filtram observações de um conjunto de dados com base na correspondência - ou não - a uma observação no outro conjunto, ou seja, são filtering joins (atuam de maneira semelhante à função filter()).
Partindo de uma base de dados cujas medidas de interesse foram selecionadas, filtradas, criadas, calculadas e unidas, podemos representá-las em gráficos, de acordo com o tipo de dado a ser representado. Com isso, no próximo capítulo, veremos como fazer gráficos a partir do pacote ggplot2, com o intuito de enxergarmos os nossos dados a partir de uma outra perspectiva.